LncPep:
http://www.shenglilabs.com/LncPep/#!/
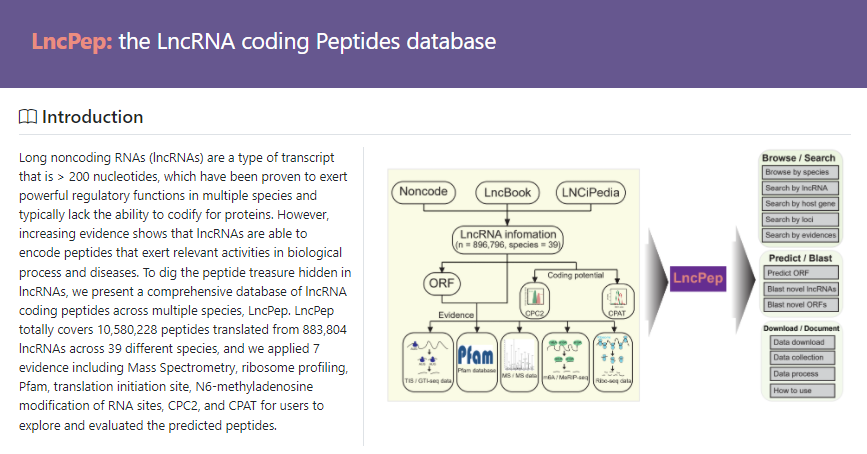
在LncPep中共涵盖 39 个不同物种的 883, 804 个 lncRNA 翻译的 10, 580, 228 个肽段。
背景数据集介绍
LncPep当中的lncRNA信息主要来自于三个数据库:
NONCODE (http://www.noncode.org/ ) ,
The LncBook database (http://bigd.big.ac.cn/lncbook )
以及LNCipedia (https://lncipedia.org ) 。
在收集到多个物种的lncRNA信息之后,作者首先基于LncExpDB (https://bigd.big.ac.cn/lncexpdb/ ) 以及CCLE数据库观察lncRNA的表达情况。
同时基于多个数据证据来证明lncRNA可以翻译成肽段。其中包括CPAT,CPC2,m6A,Pfam,Ribo-seq以及TIS六个证据来源。
除了以上的证据来源之外,作者也使用了包括
PeptideAtlas: http://www.peptideatlas.org/ ,
HPM database (https://www.humanproteomemap.org/ ),
MassIVE (https://massive.ucsd.edu/ ),
以及 PRIDE (https://www.ebi.ac.uk/pride/ )
四个质谱数据库在内的数据来作为质谱数据的证据来源。
使用
LncPep一共提供了提供了三个功能:1)数据浏览;2)数据检索以及3)数据预测
数据浏览和检索
LncPep可以直接查看各个物种当中预测到的所有可以编码肽的lncRNA信息。
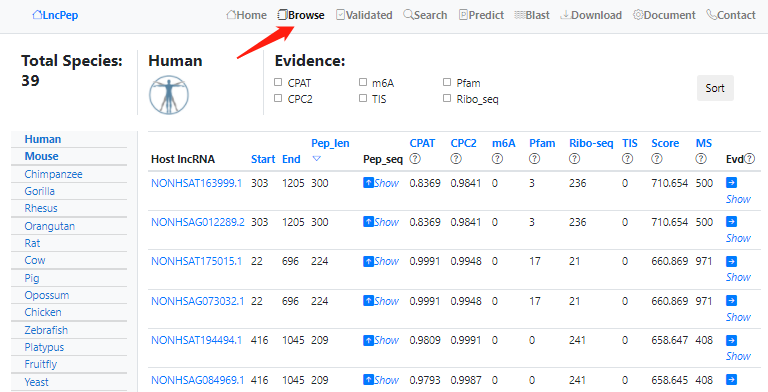
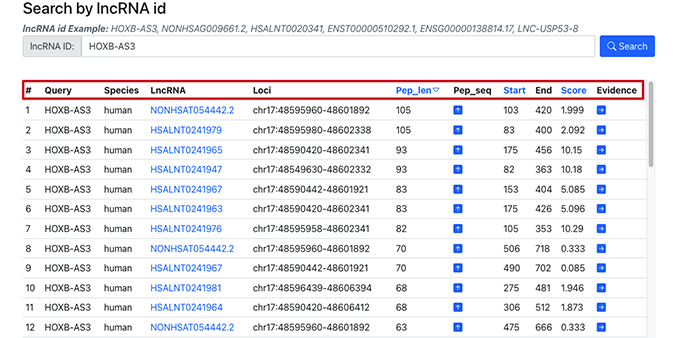
结果是以表格的形式呈现,其中点击Pep_seq可以查看编码的肽段的序列,点击Evd可以查看这个肽段是有多少个数据支持的具体信息。
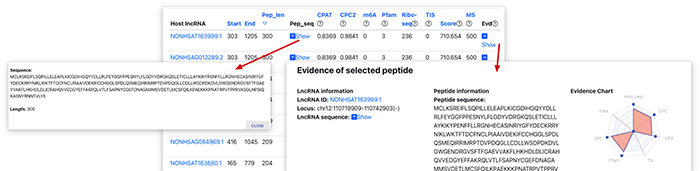
至于在检索方面,则可以基于lncRNA id, Host gene以及染色体位置等查找相关的信息。比如,我们检索HOXB-AS3
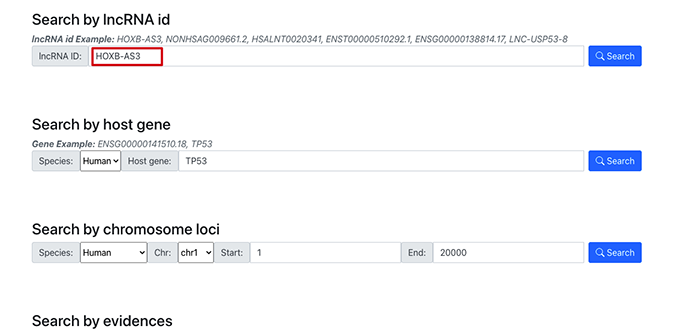
通过检索,就可以看到和这个lncRNA有关的肽段信息.
预测和blast
在预测界面,可以直接预测输入的序列的开放阅读框。同样输入的也是[[Fasta基因序列格式]]
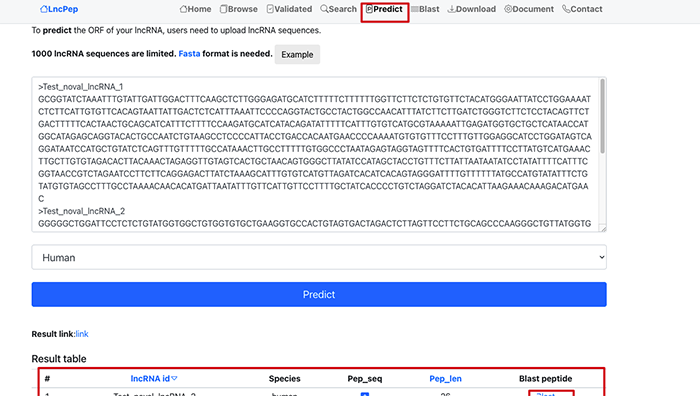
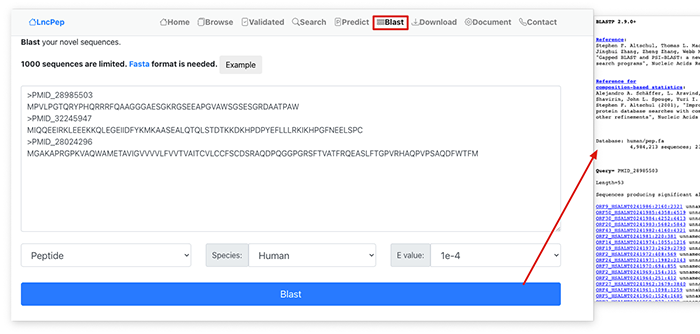
对于预测到的开放阅读框,可以直接点击Blast来比对肽段结果。Blast的结果主要是通过NCBI的BLASTP来进行分析的。
相较于SPENCER使用质谱的数据预测肿瘤有关的lncRNA肽,LncPep则是可以预测多个物种相关的lncRNA肽。同时在LncPep当中使用了多个数据来源来进行预测。相对来说预测的结果更多一些。如果是研究lncRNA的话,则可以使用LncPep来分析一下自己的目标lncRNA。














